EGFR変異型非小細胞肺がん(NSCLC)における耐性因子を特定するための新しいアプローチ
初めまして。Logbiiインターンの小林です。
今回は、ライフサイエンス×AIのテーマの3回目です。
今回は、AstraZenecaの研究チームが開発した、EGFR変異型非小細胞肺がん(NSCLC)における耐性因子を特定するための新しいアプローチについて解説していきます。この新アプローチは、ナレッジグラフをベースしたレコメンデーションシステムで、このタイプのがんに対する新たな治療法の開発につながる可能性があります。
今回はその新アプローチKnowledge graph-based recommendation framework identifies drivers of resistance in EGFR mutant non-small cell lung cancerを紹介していきます。
背景
非小細胞肺がん(NSCLC)は、肺がんの中で最も一般的なタイプで、全例の約80%を占めます。NSCLCは、肺の気泡(肺胞)を覆う細胞で発生するがんです。
EGFRは、多くの細胞の表面に存在するタンパク質で、EGFRは細胞の成長や分裂を助ける働きがあります。EGFR阻害剤は、EGFRの働きをブロックする標的治療の一種です。これにより、がん細胞の成長や分裂が止まります。EGFR阻害剤は、体の他の部位に広がったNSCLC(転移性NSCLC)や治療後に再発したNSCLC(再発性NSCLC)の治療に使用されています。
課題: 時間がかかる耐性遺伝子探し
EGFR 阻害剤 に対する耐性は、非小細胞肺がん (NSCLC) の治療において大きな障害となります。この阻害剤は、肺がんの患者に対して効果の高い薬剤と言われています。しかし、EGFR遺伝子変異が認められた非小細胞肺がんに対して、EGFR阻害剤による治療をしていると、いったんは効果が得られても、いずれEGFR阻害剤が効きにくくなってしまうこと(耐性ができること)があります。この耐性を引き起こす遺伝子を見つけるために専門家たちは多くの時間を費やさなければなりません。
一般的にその耐性を引き起こす遺伝子を見つけるためには、CRISPR-Cas9 を用いたゲノムスクリーニングを行い、数百もの耐性を引き起こしうる遺伝子を選びます。その中から、最も薬への耐性を引起こす遺伝子を研究者たちは手作業で選び、その後、疾患に関する事前知識、臨床、前臨床試験などから得られるエビデンスをもとに、どの遺伝子を優先させるか決定していきます。しかし、その作業は耐性を引き起こしうる数百もの遺伝子の中から選ぶために膨大な時間がかかってしまいます。また、研究者個人のバイアスがかかってしまう可能性もあるのです。
課題解決アプローチ
本論文のアプローチによって、上記の研究者による手作業の遺伝子の選定の時間を短縮することができます。具体的には、本アプローチにより、3,000 を超える遺伝子から 57 の耐性を引き起こす可能性の高い遺伝子が特定され、ヒットの特定にかかる時間が数か月から数分に短縮することができるのです。本論文では、3,000 を超える遺伝子から 57 の耐性を引き起こす可能性の高い遺伝子を特定するために、前臨床、臨床、文献のエビデンスを統合した異種生物医学知識を学習したレコメンデーションシステムを構築しました。このシステムは、EGFRi耐性の潜在的なメカニズムに関連する様々なタイプのエビデンス間(臨床や文献など)のトレードオフに基づいています。それによって、耐性を最も引き起こす可能性の高い遺伝子をランク付けをしていくのです。
CRISPR スクリーニングとその課題
非小細胞肺がんを治療するために投与するEGFR阻害剤は、投与し続けるうちにその阻害剤に対して耐性ができてしまう可能性があります。その耐性を引き起こす遺伝子を見つけ、その遺伝子を壊さなければなりません。そのために使用されるのが、CRISPRスクリーニングです。CRISPR スクリーニングとは、CRISPR-Cas9というゲノム編集技術の一種を用いて、ゲノムワイドノックアウト、ノックダウン、ノックインを行うことを指します。具体的には、CRISPR-Cas9はDNA二本鎖を切断してゲノム配列の任意の場所を削除、置換、挿入することができます。ゲノムワイドノックアウト( genome-wide knock out)とは、その削除にあたります。ノックダウン(knock down)とはノックアウトとは異なり、遺伝子の機能を大きく減弱させるものの完全には失わせないことを指し、ノックイン(knock-in)とは、削除するだけでなく新たな配列を挿入することをいいます。つまり、CRISPRスクリーニングは任意の遺伝子の配列を変えることでできるハサミみたいなものです。このCRISPRスクリーニング(ハサミ)を用いて、薬剤耐性を引き起こす遺伝子を切断できるのです。
しかし、 薬剤耐性を引き起こす遺伝子の切断の方法はわかったものの、課題はその遺伝子を見つけるのに時間がかかるということです。生物学的に妥当性の高い耐性遺伝子に絞り込むために、研究者は手作業でトリアージとバリデーションを行わなければなりません。このプロセスは疾患に関するナレッジ、臨床や前臨床試験から得られるエビデンスを集約し、検証のためにどの遺伝子を優先させるかを決定するため、時間がかかります。また、深い専門的な知識に依存しているため、結果が研究者個人のバイアスに左右される可能性があります。本論文の目的は、このような手作業のトリアージを、なんとかレコメンデーションシステムを用いて、短縮できないかというものです。多様なエビデンス(ナレッジ、臨床や前臨床試験)を効率的に統合し、薬剤耐性をもたらす最も有望な候補遺伝子を同定することを本論文は目指します。
解決策:レコメンデーションシステムの概要
これまでは、様々なエビデンスと照らし合わせて手作業で耐性を引き起こす遺伝子を絞り込んでいました。しかし、その手作業で行っていた時間をどのように短縮するのでしょうか。本論文では、多目的最適化(multi-objective optimization)を用いたレコメンデーションシステムを使ってその時間を短縮することを実現させました。
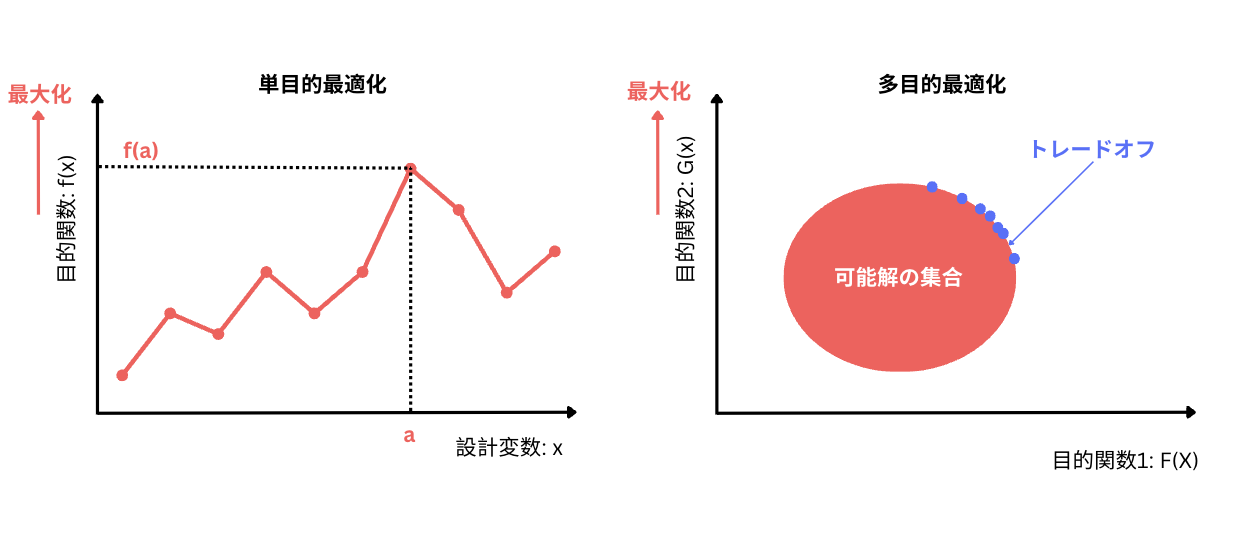
では、多目的最適化とはなんでしょうか。多目的最適化とは、目的関数が複数ある最適化のことを指します。逆に、目的関数が「1つ」だけの最適化は単目的最適化といいます。図1は、それぞれ単目的最適化と多目的最適化を表しています。
左の図のように単目的最適化における最適解は1つです。しかし、多目的最適化の最適解は、右の図の紫の点のように複数あります。複数の目的関数はトレードオフの関係にあります。目的関数1を大きくすると、目的関数2が小さくなってしまうのです。たとえば、ある性能を改善しようとすると、ほかの性能が悪化するような関係にある場合が多く存在します。右の図の円周上に、最適解があり、この紫の点の最適解の集合のことを「パレート最適解」と呼ぶのです。本論文で使用する多目的最適化というのは、このパレート最適解の集合を求めることを意味しています。
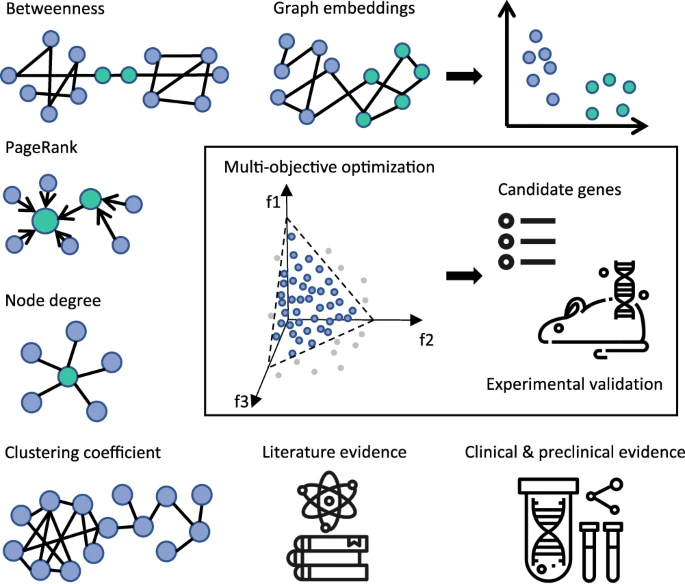
本論文では、図2にある「Betweenness」「Graph embeddings」「PageRank」「Node degree」「Clustering coefficient」「Literature evidence」「Clinical & preclinical evidence」を目的関数として使用します。つまり、これらの目的関数のパレート最適解の集合を求めることによって、耐性を引き起こす遺伝子を絞り込むのです。
多目的最適化で使った特徴量
前提として多目的最適化を行う際に、多くの特徴量が必要になります。特徴量が多いと解の候補をより細かく評価し、より最適な解を選択できるからです。多目的最適化は、複数の目的関数を同時に最小化または最大化することを目的としています。解の候補を評価するためには、目的関数の値以外にも、その解の候補が満たすべき条件や限制などの情報を考慮する必要があります。例えば、工学設計において、コストと重量、強度と耐久性など、複数の目的関数を満たすことが求められます。しかしそれ以外に材料、環境条件、安全性などの情報を考慮し、多くの情報を扱うことで、最適な解を選択できます。
本論文では、最適解を探すために27の特徴量が選ばれました。
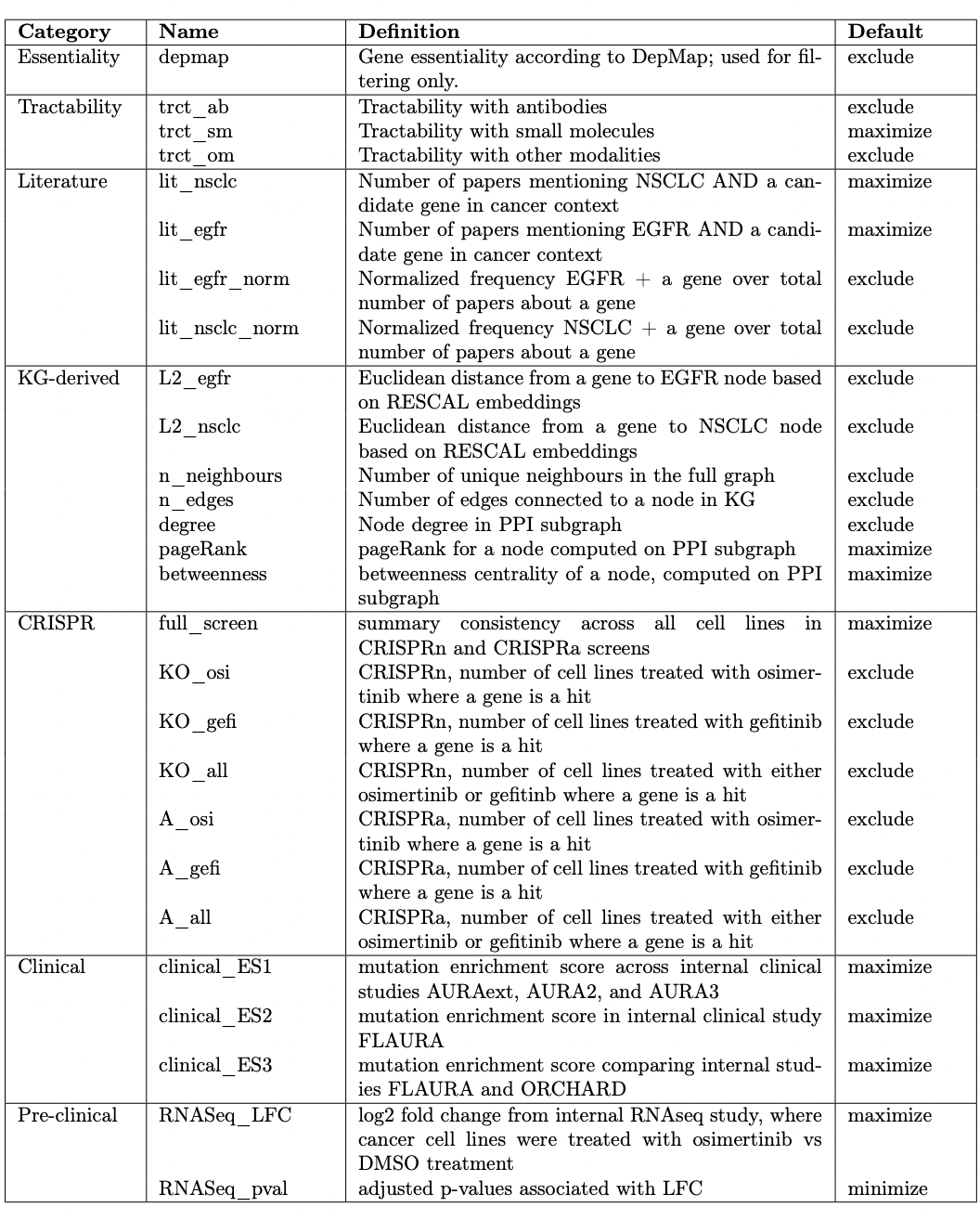
表:27の特徴量
表は、その27のそれぞれの特徴量を示しています。大きくカテゴライズすると、7つの特徴量に分類されます。Defaultの列における「exclude」は、最適化プロセスに含むべきではないという結果出た特徴量という意味です。多目的最適化のおいて、多くの特徴量を最初扱いますが、それらすべてが重要であるわけではありません。一部の特徴量は、最適化プロセスから除外され、問題を単純化し、計算上効率的にします。特徴量の除外は、関連性、重要性、その他の特徴量との相関などの基準に基づいて行うことができ、表の「exclude」とある特徴量は使用されない特徴量のことを指します。また、maximizeまたはminimizeと示しているものは、最適化の方向を表し、最適化プロセスとして使用されるものです。
この表は、「exclude」かどうか判断するための判断材料の一つとして各特徴量がどのくらい相関しているか示したものです。ここから2つのことが読み取れます。1つ目は、graphのカテゴリーが強く相関していることです。前の節で説明したpagerankやbetweennessなどがgraphのカテゴリーです。つまり、多目的最適化においてこの特徴量は外せないものと言えます。2つ目は、CRISPRのカテゴリーはマイナスに相関していることです。紫の部分がそのマイナスに相関している部分に当たります。
つまり、多目的最適化の精度をあげるために、図2の他に様々な特徴量をあげ、合計27の特徴量を多目的最適化プロセスに必要かどうか分析しました。それぞれの相関係数を調べ、「exclude」か「maximize/minimize」を決定し、まとめたものがこの表です。
SkywalkR インタラクティブ インターフェイス
図3と図4は、SkywalkRの対話型インターフェースであり、ユーザーはCRISPRヒットの再ランク付けを、様々な目的の組み合わせに基づいて行うことができます。
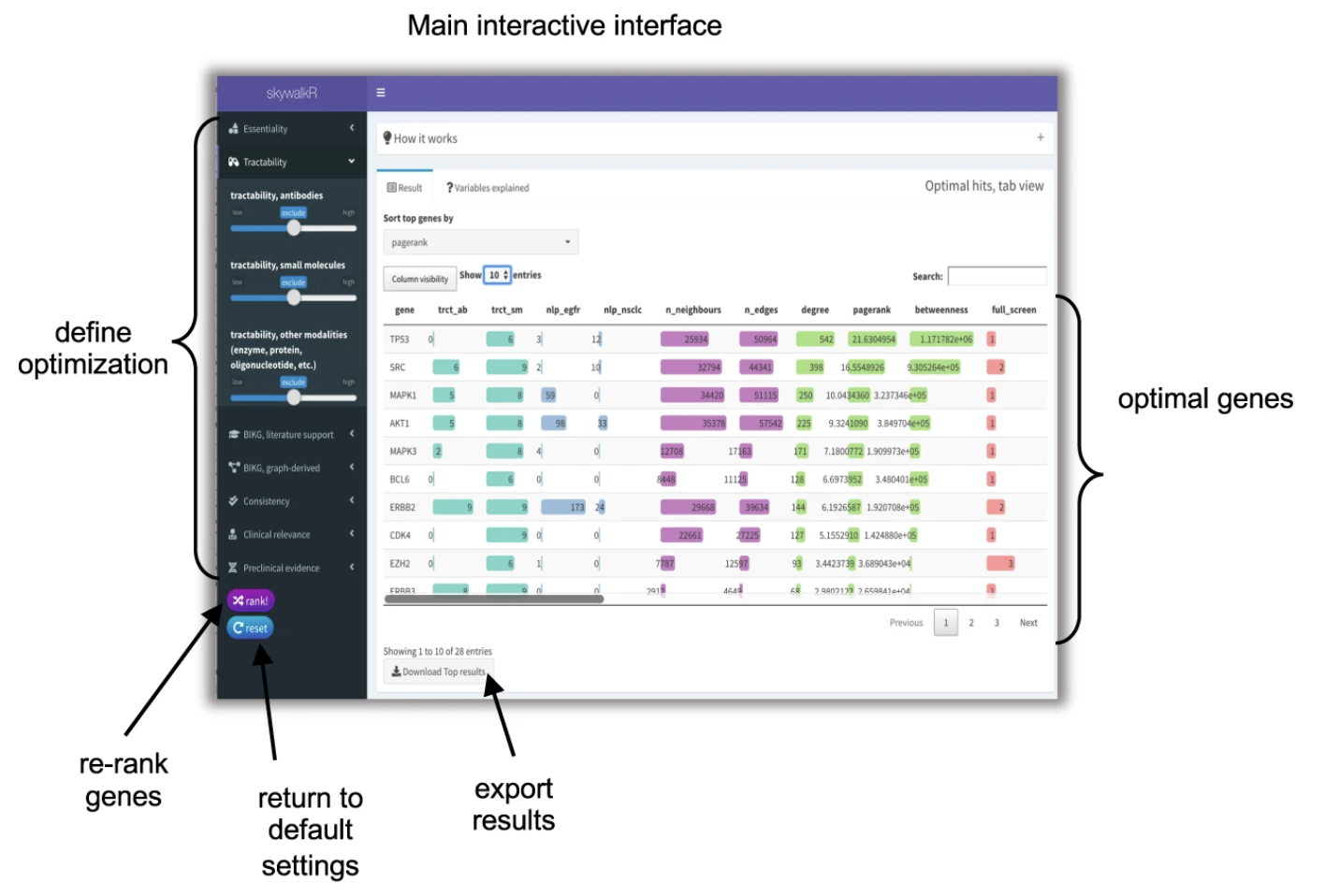
図3:メインのインターフェース
図3のサイドバーパネルには、EGFRi耐性の遺伝子推奨を最適化するために使用できる目的のリストが表示されています。各目的はスライダーで表され、ユーザーは最適化に含める目的を選択できます。また、ユーザーは最適化の方向を「最小化」または「最大化」で指定できます。
例えば、ユーザーがEGFRi耐性と高い関連性がある遺伝子を特定したい場合、目的の「EGFRi耐性」を選択し、最適化の方向を「最大化」に設定できます。これにより、EGFRi耐性と高い関連性がある遺伝子のリストが右に表示されます。
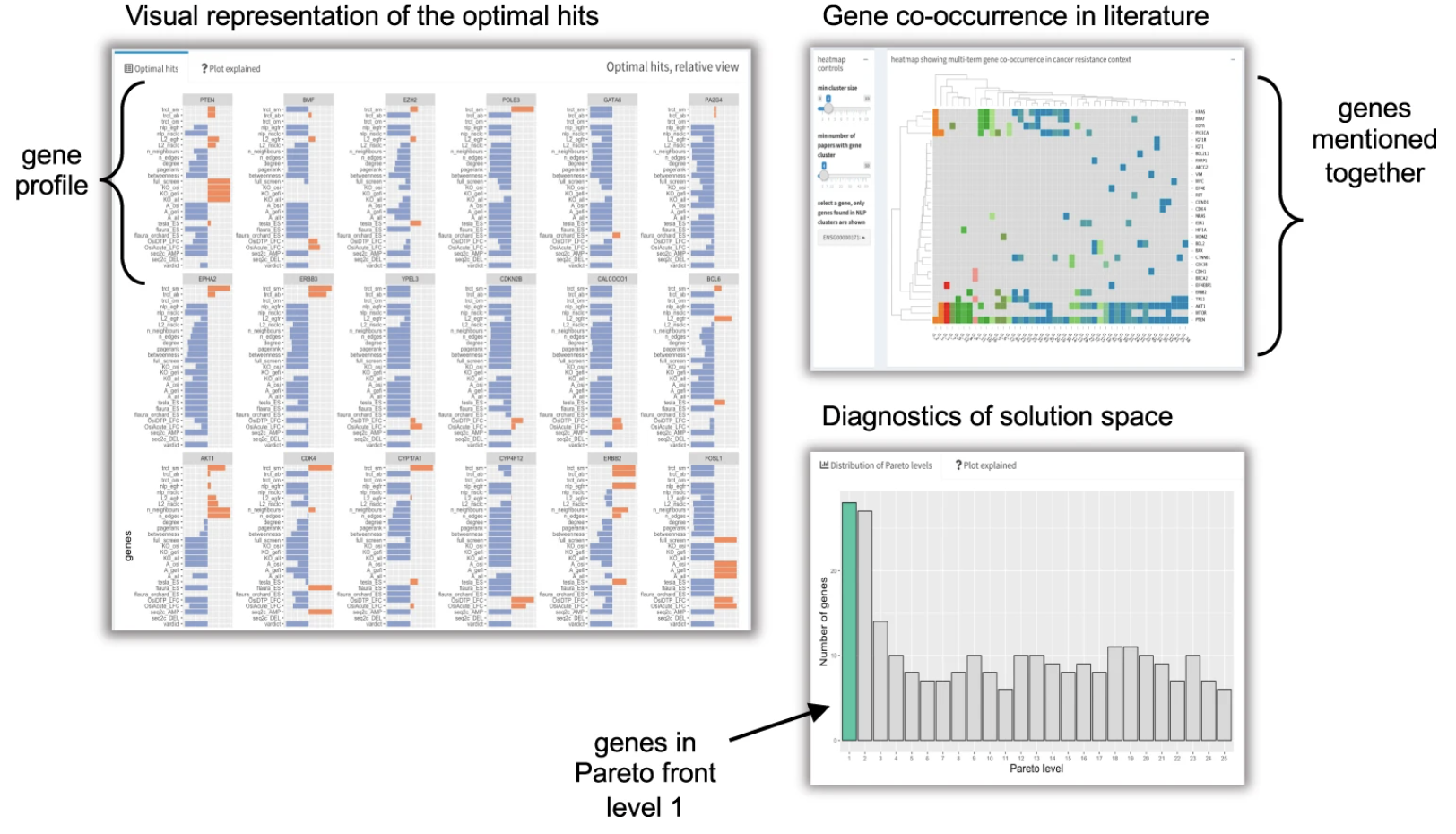
図4:サブインターフェース
図4のサブインターフェースは、最適化の結果を調査するために使用されます。
相対ビュー(Relative view):図4の中の左の図は、相対ビューといいます。この相対ビューは、レコメンドされる遺伝子のプロファイルを表示し、各遺伝子は線で表されます。これらの線は、各遺伝子に対するさまざまな目的の値を示しています。これにより、遺伝子を比較し、複数の目的で高いパフォーマンスを発揮する遺伝子を特定できます。 例えば、EGFRi耐性と高い関連性があり、複数の証拠タイプによって支持される遺伝子を探し出すとしましょう。相対ビューでは、「EGFRi耐性」目的と「複数の証拠」目的の両方で高い値を持つ遺伝子を探し、画面上に表示します。
共起ヒートマップ(Co-occurrence heatmap):図4の右上のヒートマップは、共起ヒートマップと呼ばれます。これは、EGFRi耐性の文脈で一緒に言及されることが多い遺伝子のクラスターを示しています。これにより、関連する遺伝子を特定し、同じ生物学的経路に関与している可能性がある遺伝子を見つけることができます。 例えば、EGFRシグナル伝達経路に関与する遺伝子を特定する場合、共起ヒートマップでは、一緒に言及されることが多い遺伝子のクラスターを探します。これらの遺伝子のクラスターは、同じ生物学的経路に関与している可能性が高いを意味します。
棒グラフ:最後に、図4の右下のグラフについて説明していきます。この棒グラフは、上位の推奨遺伝子に対して目的ごとに標準化された値を示しています。これにより、遺伝子が各目的でどのように互いに比較されるかを確認できます。 例えば、EGFRi耐性と最も関連性が高い上位10の遺伝子を特定する場合、棒グラフでは、「EGFRi耐性」目的で最も高い値を持つ遺伝子を探し出し、表示させます。
このように、SkywalkRのインターフェースは、専門家が異なる目的に基づいてレコメンドがどのように変化するかを見ることができる貴重なツールであり、NSCLCの治療や薬剤開発に役立つ情報を提供することができます。
考察
本研究の発見は重要であり、ナレッジグラフを用いてEGFR変異型NSCLCの新たな治療標的を特定することができる可能性を示しています。これは、このタイプのがんの新たな治療法の開発において重要なステップです。
研究者たちは現在、特定した遺伝子のさらなる検証と、これらの遺伝子を標的とする新薬の開発に取り組んでいます。これは、がん研究の分野において刺激的な発展です。EGFR変異型NSCLCの新たな治療法の開発は緊急に必要とされており、この研究は、このタイプのがん患者に役立つ新薬の開発につながる可能性があります。
本ブログを読んでいただき、ありがとうございます!より詳しい情報や研究の詳細に興味を持たれた方は、オリジナル論文をぜひ一読してみてください。