初めまして。Logbiiインターンの高橋です。
今回は、ライフサイエンス×AIのテーマの2回目です。
現在の生物学においては様々な論文などで溢れかえっていますが、生物医学用語はとても曖昧で、1つのフレーズが論文を書く科学者によって意味が異なってしまう場合があります。例えば変異ヘモグロビンα2は遺伝子かタンパク質を指します。さらに論文の著者がタンパク質Aとし、機械学習などに用いるアノテーションを遺伝子Aとした場合、アノテーションの有用性が低くなってしまいます。これを解決するべく、この論文では生物医学のための先進的なアノテーションツール、NERO: a biomedical named-entity (recognition) ontology with a large, annotated corpus reveals meaningful associations through text embedding[1]を開発したとのことです。
今回はこのNEROという論文のピックアップをして紹介していきます。
概要
NEROは以下の6つの要素で構成されています:
- 分子生物学、遺伝学、生化学、医学における、新しい固有表現抽出のためのオントロジー(Named Entity Recognition Ontology:NERO)
- アノテーターへのガイドライン
- 固有表現の絵文字表記
- 190,679の固有表現と2つ以上の表現を繋ぐ43,438のイベントをカプセル化
- 検証した固有表現抽出(Named Entity Recognition : NER)モデル
- このコーパスを用いた生物医学的な関連性を示す埋め込みモデル
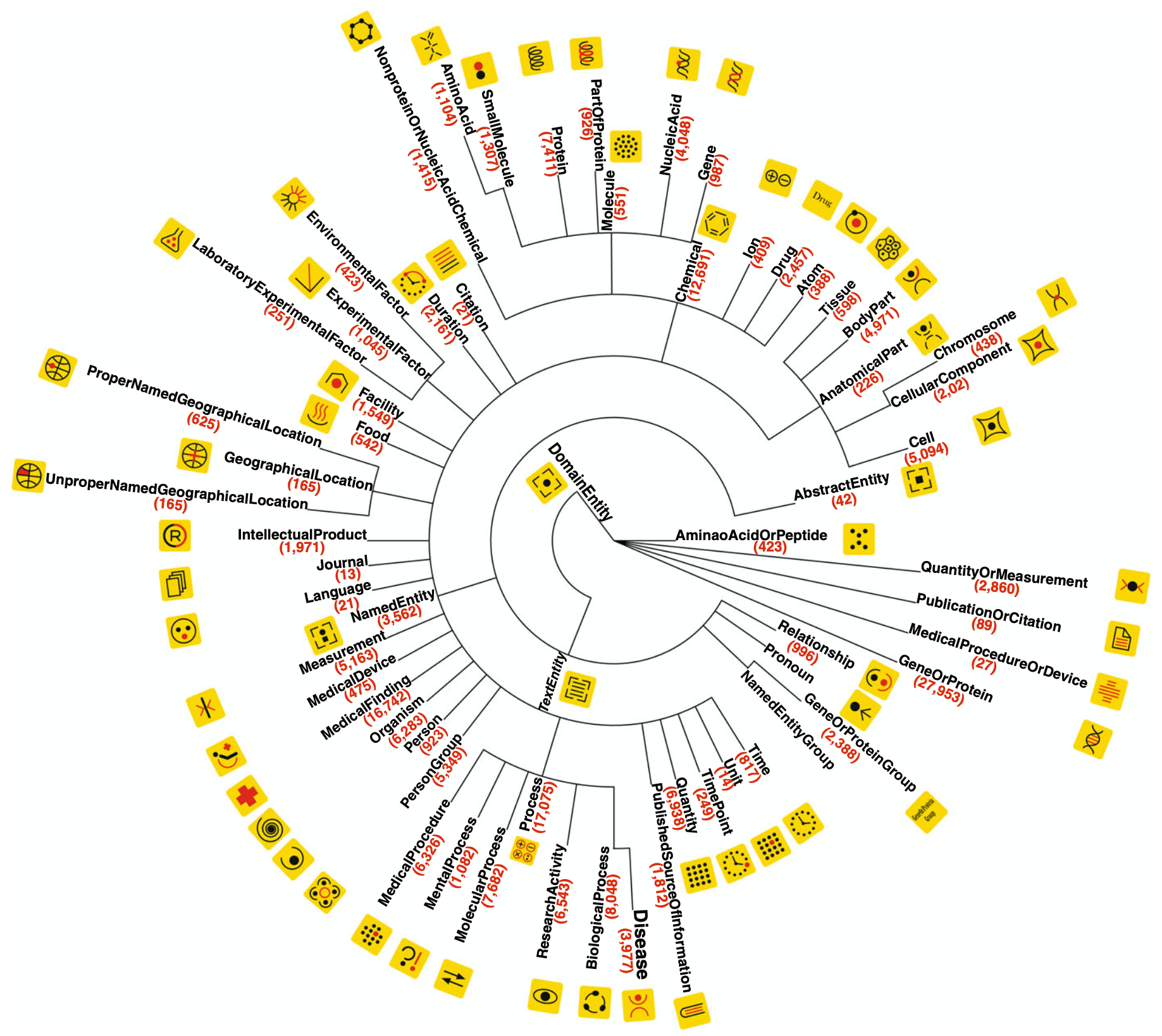
図1 NEROのイメージ図
NEROのイメージを表しているのが図1です。
この図では、多分木ツリーとしてオントロジーが示されています。分類ノードはオントロジーの各クラスに対応しています。各クラスの下の括弧は、コーパス内のクラス言及数を表しています。NEROでは35,865文のアノテーションを行い、うち190,679は固有表現、43,438は2つ以上の表現を繋ぐイベントで構成されています。
“DomainEntity“のクラスターの下には”AnatomicalPart“, “Chemical“, “Process“の代表的なクラスがあり、全ての固有表現の半分以上がこの3つのクラスに由来しています。また、各クラスにおいて、コーパスを使用する際に手動でアノテーションを付ける事を簡略化し、さらにビジュアル的にもわかりやすいようにそれぞれの要素を表す絵文字が用意されています。例えば、”Gene“であれば遺伝子の絵、”BodyPart“であれば体の一部分を示しています。
コーパスを作る際にポスドク・生物医学業界の専門家たちのチームを採用することにより、大規模な生物医学コーパスにアノテーションをつけて、幅広い自然言語処理と生物医学の機械学習タスクを可能にしています。
NEROにおいてのアノテーションは、”GeneOrProtein“のような曖昧な概念を”Gene“と”Protein“で分離し、これらのいずれかもしくは両方に対応するというパターンを利用して、アノテーションに関する適切な曖昧さを表現し、テキストの不確実さを保持します。
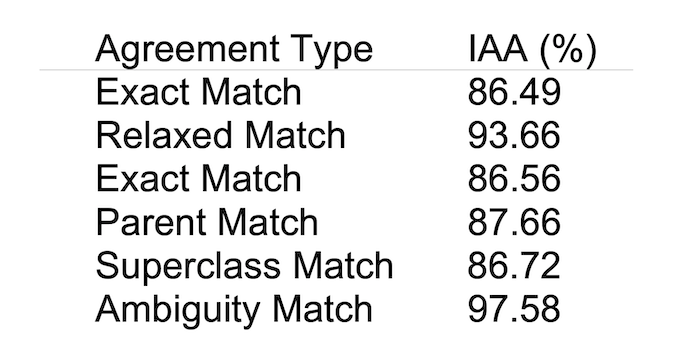
また、アノテーションされた35,865文のうち8,650文は、非常に高いアノテーターたちの合意によって付けられており、合意が取れている割合は表1のようになっています。
IAAとはInter-annotator Agreementと言い、アノテーションをするアノテーターの間で合意が取れている割合を表しています。
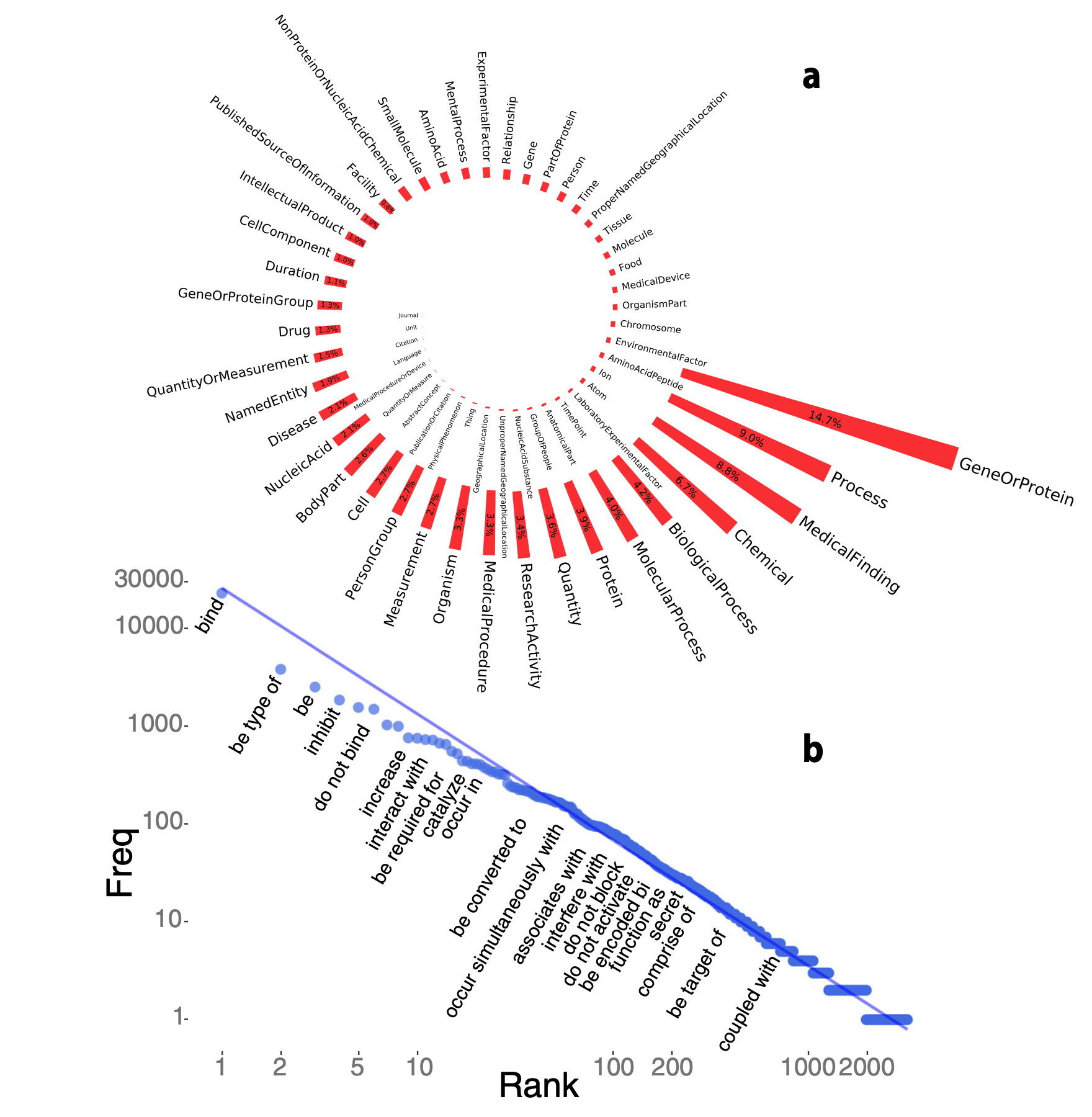
NERO内の固有表現の出現頻度は以下の図2のようになっています。
図2 NEROに含まれる固有表現の出現頻度
これらを元に、アノテーション付きコーパスを使用したい研究者向けに、NERO-nlpというパッケージ[2]も開発しています。
実験内容
NEROの実用的なアプリケーションとして、以下の2つを提案・実験しています。
-
固有表現を識別する機械学習モデル
-
単語の埋め込み
・実験1 機械学習モデル
まず初めにNERsuite[3]を使用しデータセットを教師データ・テストデータに分割し、交差検証をしました。分類結果は表2のようになりました。全体的な固有表現抽出のパフォーマンスは中程度で、適合率は54.9%、再現率は37.3%、F値は43.4%となっています。最も数値が高かった”GeneOrProtein“は、ベースラインで適合率は67.0%、再現率は65.3%、F値は66.2%を出しました。また、NERsuiteのデフォルトのベースライン実装に加え、さらに精度を向上させるために、専門用語の辞書を参照する辞書機能を追加しました。その結果、適合率は54.7%、再現率は37.9%、F値は43.8%となり、辞書機能を追加してもF値の向上は0.35%に留まりました。次に、NERsuiteをベースモデルとして、スタッキングと呼ばれるアンサンブル手法を実装しました。その結果、ベースライン結果と比較してF値が0.27%増加しました。
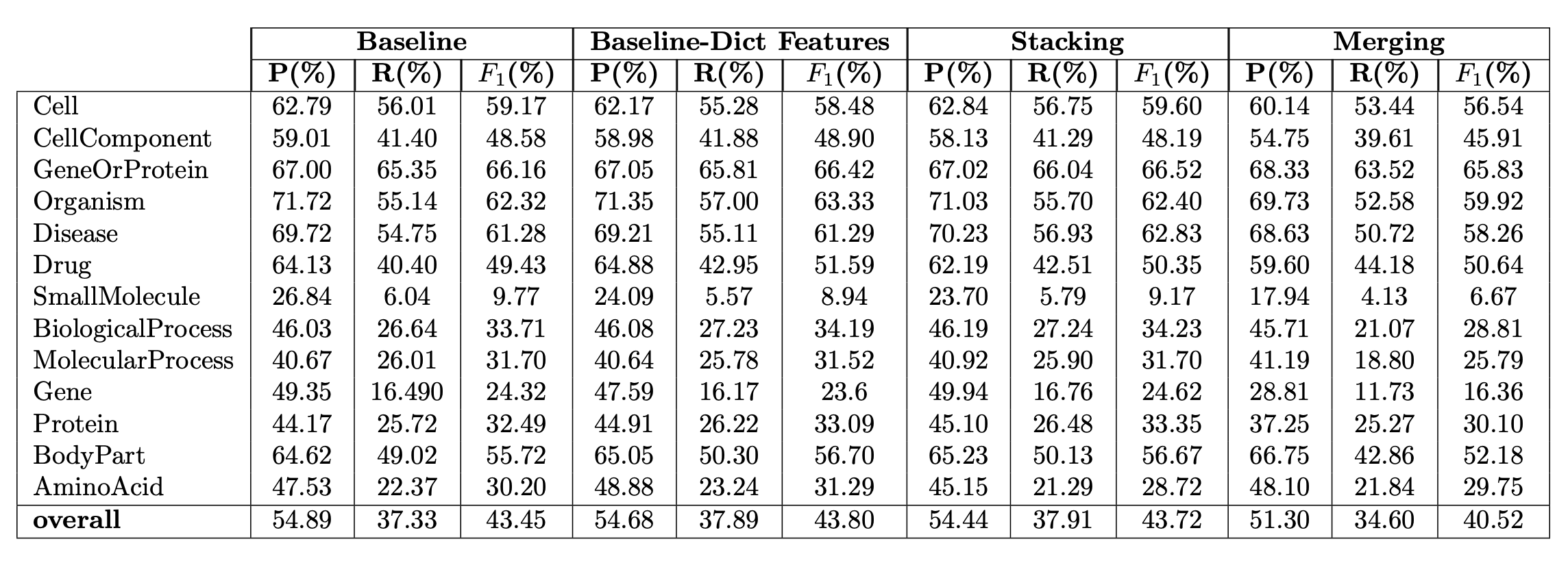
表2 データセットの10%で評価された固有表現抽出の実験結果
・実験2 単語埋め込み
この実験では単語埋め込みモデルとしてword2vec[5]を使用し、NEROのアノテーション付きコーパスの埋め込まれた意味を構築し、疾患や薬剤に関するエビデンスと比較しました。使用したコーパス(ウィキペディア、Elsevierの記事、ロイターの記事)について、疾患や薬剤に関連する固有表現を300次元の空間に埋め込みました。今回はgensim[6]というパッケージを用いてword2vecを実装しています。
NEROに基づいて単語埋め込みを評価するために、2つの疾患の観点(「重症度」と「性別」)、2つの薬剤の観点(「毒性」と「費用」)について分類しました。
まず疾患について、ここでは以下のように対称的な用語のペアを使用して、疾患の重度・軽度の軸を構築しました。
- “harmful”, “beneficial”
- “serious”, “benign”
- “life-altering”, “common”
- “disruptive”, “undisruptive”
- “dying”, “recovering”
- “dangerous”, “safe”
- “threatening”, “low-priority”
- “high mortality”, “harmless”
- “costly”, “cheap”
- “hospitalized”, “self-administered”
- “hospital”, “work”
- “debt”, “savings”
- “low quality of life”, “undisruptive”
- “hazard”, “routine”
また、この軸での疾患の埋め込みを、各疾患との生活の負担に関するWHOのデータ(DALYs13)[7]と比較しました。その結果、0.329 (p=0.0614, n=33)の相関関係を見つけることが出来ました。
同様にして、性別における対称的なペアを使用し、性別の軸を以下のように構築しました。
- “male”, “female”
- “prostate”, “ovary”
- “penile”, “uterine”
- “penis”, “uterus”
- “man”, “woman”
- “men”, “women”
- “masculine”, “feminine”
- “he”, “she”
- “him”, “her”
- “his”, “hers”
- “boy”, “girl”
- “boys”, “girls”
この軸での疾患の埋め込みを、2003年から2011年までのアメリカの保険記録を用いて、男性と女性の疾患の有病率と比較しました。この結果、0.436 (p=1.46×10−13, n=261)の相関が見つかりました。
次に薬剤について、以下の対称的な用語のペアを使用して、薬剤の毒性の軸を構築しました。
- “harmful”, “beneficial”
- “toxic”, “nontoxic”
- “noxious”, “benign”
薬剤の毒性の軸での疾患の埋め込みを、薬剤固有の半数致死量(LD50データベース[8]に記載されているモデル動物の50%致死量)と比較しました。その結果、0.32(p=1.1×10-4)の相関関係を見つけることが出来ました。
同様にして、費用における対称的なペアを使用し、費用の軸を以下のように構築しました。
- “expensive”, “inexpensive”
- “costly”, “cheap”
- “brand”, “generic”
- “patented”, “off-patent”
薬剤の費用の軸での疾患の埋め込みを、IBM MarketScanデータベース[9]と比較しました。その結果、費用の軸と各薬剤の実際の価格への薬剤予測の相関は0.42(p=1.5×10-15)でした。
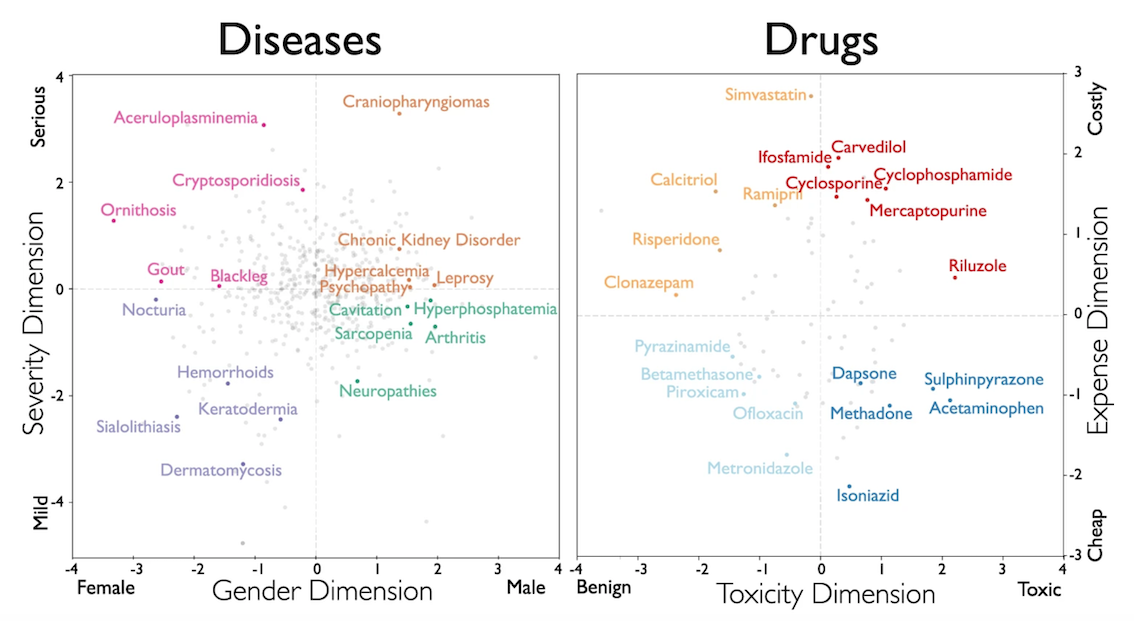
疾患の観点(「重症度」と「性別」)、2つの薬剤の観点(「毒性」と「費用」)での埋め込みを表すのが、図4になります。
図4 疾患と薬剤の二次元投影
オウム病など、疾患が性別の軸で低くなると、男性よりも女性を苦しめる可能性が高くなります。ハンセン病のように、疾患の重症度の軸で高く突き出た場合、かなりの苦痛を被る可能性があります。毒性の軸で負の値の薬は、より深刻な副作用と関連する傾向があります。たとえば、筋萎縮性側索硬化症の治療薬であるリルゾールは、異常な出血から吐き気や嘔吐に至るまでの副作用があります。費用の軸で値が高い薬剤は、特許を失う前は多くの費用がかかっていたシンバスタチンのように、厳しい医療費を表します。これらの結果の頑健性は、科学的コーパスが、追跡調査に値する仮説の自動生成に使用できることを示唆しています。
議論
この研究の主な制限として、エンティティの分類におけるすべてのレベルの粒度をカバーしなかったこと、オントロジークラスの頻度がヘビーテイルで分布しているため、いくつかの概念タイプが十分に表現されていないことなどが挙げられています。
参考文献
[3] Labelling Sequential Data in Natural Language Processing
[4] Spacy・Industrial-Strength Natural Laguage Processing
[5] word2vec: Distributed Representations of Words and Phrases and their Compositionality
[6] Gensim: Topic modelling for humans
[7] History of global burden of disease assessment at the World Health Organization
[8] ChemIDplus Help – A structure searchable database of 400,000+ chemical substance records
[9] IBM MarketScan Research Databases for life sciences researchers