今回は、退院サマリーから薬剤有害反応(Adverse Drug Reaction:ADR)の自動抽出を行なった論文「Developing A Deep Learning Natural Language Processing Algorithm For Automated Reporting Of Adverse Drug Reactions」[1]をピックアップして紹介していきます。
論文の概要
ADRの検出は、薬剤の安全性とリスク・ベネフィット・プロファイルを理解する上で非常に重要です。ADR は患者の罹患率において重大な原因であり、世界中の急性期病院入院患者の 5-10%を占めています。ADR の自発的な報告は長い間、標準的な報告方法でしたが、この方法は過少報告の割合が高いことが知られており、ファーマコビジランスの取り組みを制限する問題になっています。ADR 報告の自動化は、報告率を高めるための代替手段となりますが、これは他の薬剤に関連する有害事象の過剰報告につながるリスクがあります。
著者は、大学病院における退院サマリーで ADR を識別するために、深層学習の自然言語処理アルゴリズムを開発しました。そのモデルは 2 段階で開発されました。まず、学習済みモデル(DeBERTa[2])を、ラベル付けされていない 15 万件の退院サマリーでさらに事前学習し、アノテーション付き退院サマリー 861 件のコーパスで ADR の言及を検出するために追加学習しました。このアルゴリズムが ADR と他の薬剤に由来する有害事象を確実に区別できるように、アノテーション付きコーパスには、検証済みの ADR レポートと、交絡する他の薬剤に由来する有害事象の両方を準備しました。最終的なモデルは、ADR に関する記述を含む退院サマリーを識別するタスクに対して、ROC-AUC が 0.934(95%CI 0.931 – 0.955)と良好な性能を示しました。
本研究は、ADR の自動検出のために開発された自然言語処理(Natural Language Processing:NLP)モデルの可能性を示しています。このアプローチは、現在の手法の報告不足を解決し、現在の臨床ワークフローのリソースの制限を回避し、病院内の ADR 報告率を向上させることができます。著者らの医療ネットワークに特有の電子医療記録(Electronic Medical Records:EMR)データで追加学習することで、モデルは退院サマリーのフォーマットのパターンを学習することができ、様々なクラスに分類できるようになりました。
NLP を用いた ADR 検出
・研究目的
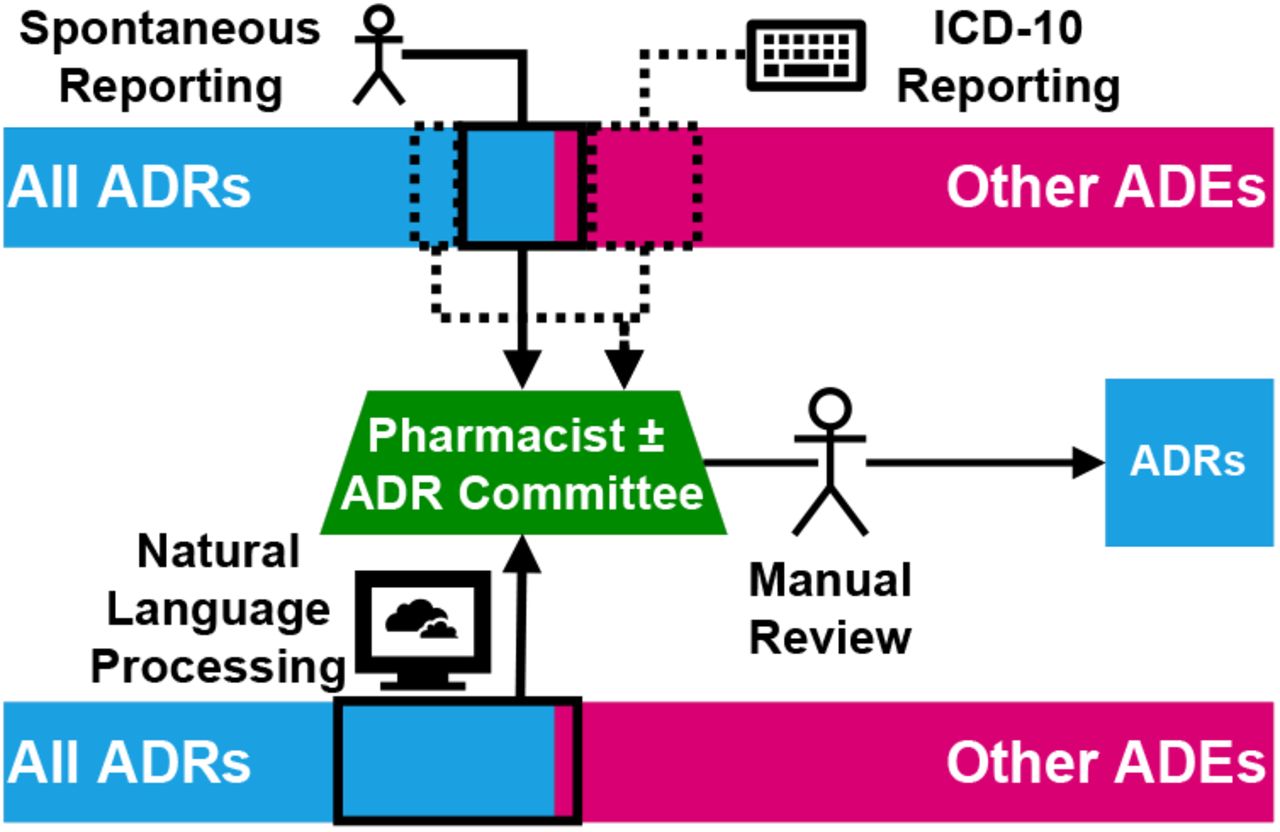
本研究の目的は、退院サマリーにおける ADR を特定するための NLP アルゴリズムを開発し、ADRを他の薬剤有害事象(Adverse Drug Events:ADE)から区別してADR 報告を補強することです(図 1 参照)。そのため、ADR と ADE の両方についてアノテーションした新しいコーパスを開発しました。著者らはBERT[3]やBioBERT[4]のような、事前に学習した巨大な言語モデル(Large Language Models:LLM)を使用しました。国(オーストラリア)および業界の文書作成慣習と語彙に適応するために、著者らの大学病院からアノテーションなしの退院サマリーの大規模コーパスでモデルを事前学習した上で、上記のアノテーション付きコーパスで追加学習をしました。ADR と ADE の両方に特化したデータを使用することでこの目的を達成します。
図1 ADR報告の流れ
[下]NLPアルゴリズムを用いたADR報告
・研究データ
オーストラリア・メルボルンにある 900 床の大学病院の臨床研究データウェアハウスから、5 年間(2015 ~ 2020 年)にわたる 861 件の退院サマリーを収集しました。すべての退院サマリーは、Y40-59 ICD-10 コード(ADE コード)でコード化された入院患者からのものであり、これは「すべての入院患者に ADE が生じていた」と臨床コーダーによって評価されたことを意味しています。この結果には、著者らの施設の ADR 委員会によって事前に審査・検証された 231 件の真の ADR も含まれていました。入院患者の診療科は多岐に渡っていました(一般内科 23%、老年科 10%、外科 15%)。
検証方法
・アノテーション
退院サマリーの薬剤名と既知の副作用のアノテーションには、アノテーションツール「Prodigy[5]」を使用しました。初めに、最初のアノテーション付きコーパスを手動で作成しました。その後、オープンソースのMed7[6]モデルを用いて、以降のすべてのバッチ(バッチあたり 100-200 テキスト)でアクティブラーニングを実施しました。このプロセスを、すべての文書にアノテーションが施されるまで繰り返しました。ラベルは「DRUG」と「ADR」の 2 種類を用いて、NER トレーニングデータを作成しました。1 人のアノテーターがすべてのアノテーションを行い、上級臨床医がレビューしました。
・処理とモデル学習
著者は、多くの類似した NLP タスクにおいてBERT[3]よりも優れた結果を示すDeBERTa[2]を使用しました。事前学習済みバージョンは、HuggingFace Transformers library[7]から入手可能です。臨床テキストを用いた事前学習は、EMR から 15 万件のアノテーションのない退院サマリーのコーパスを用いて、マスク言語モデリング(Masked Language Modeling:MLM)で行いました。このステップでは、未アノテーションコーパスの 15%のトークンがマスクされ、モデルは周囲のテキストからマスクを予測するように学習しました。
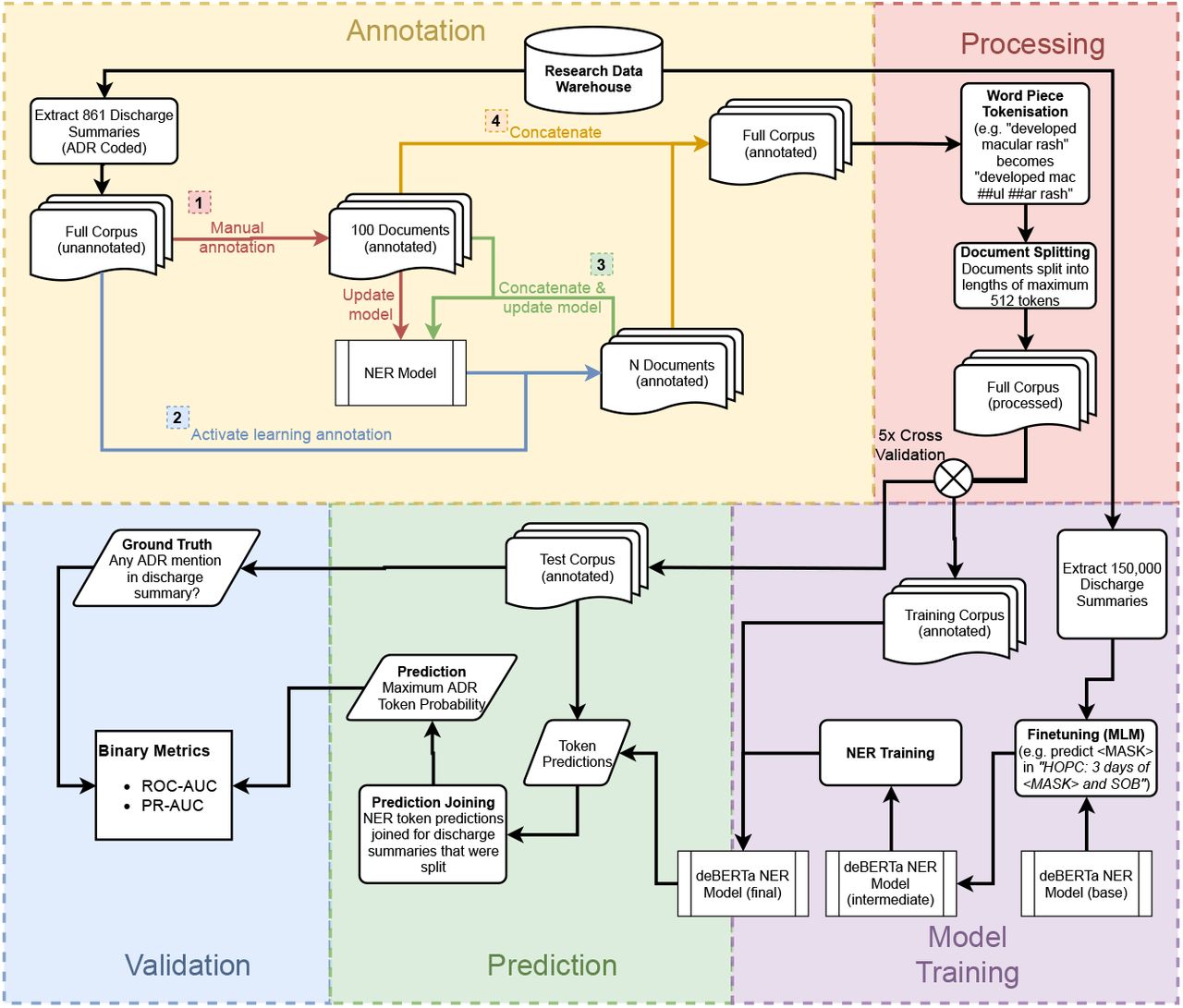
アノテーション付き文書は WordPiece トークナイザーを用いてトークン化しました。これらのトークン化された文書を、薬剤および ADR の NER タスクのために追加学習しました(図 2 参照)。最大確率の NER 単語ラベルを、退院サマリーが ADR を含むか否かの分類に使用し、最終モデルは K-分割交差検証を用いて文書レベルで評価しました。
検証環境としては、Python 3.9.6 と Nvidia 1080ti を用いました。すべてのコードはhttps://github.com/AustinMOS/adr-nlpで入手可能です。
図2 モデルのトレーニングと評価プロセス
アノテーションは4つのステップに分割されます。「Annotation」の ステップ2(Active learning annotation) と ステップ3(Concatenate & update model)は、毎回 N個の退院サマリーで順番に繰り返されます。全データのアノテーションが終わるか、更新された NER モデルの改善が最小限になった時点でこのプロセスを終了します。
結果
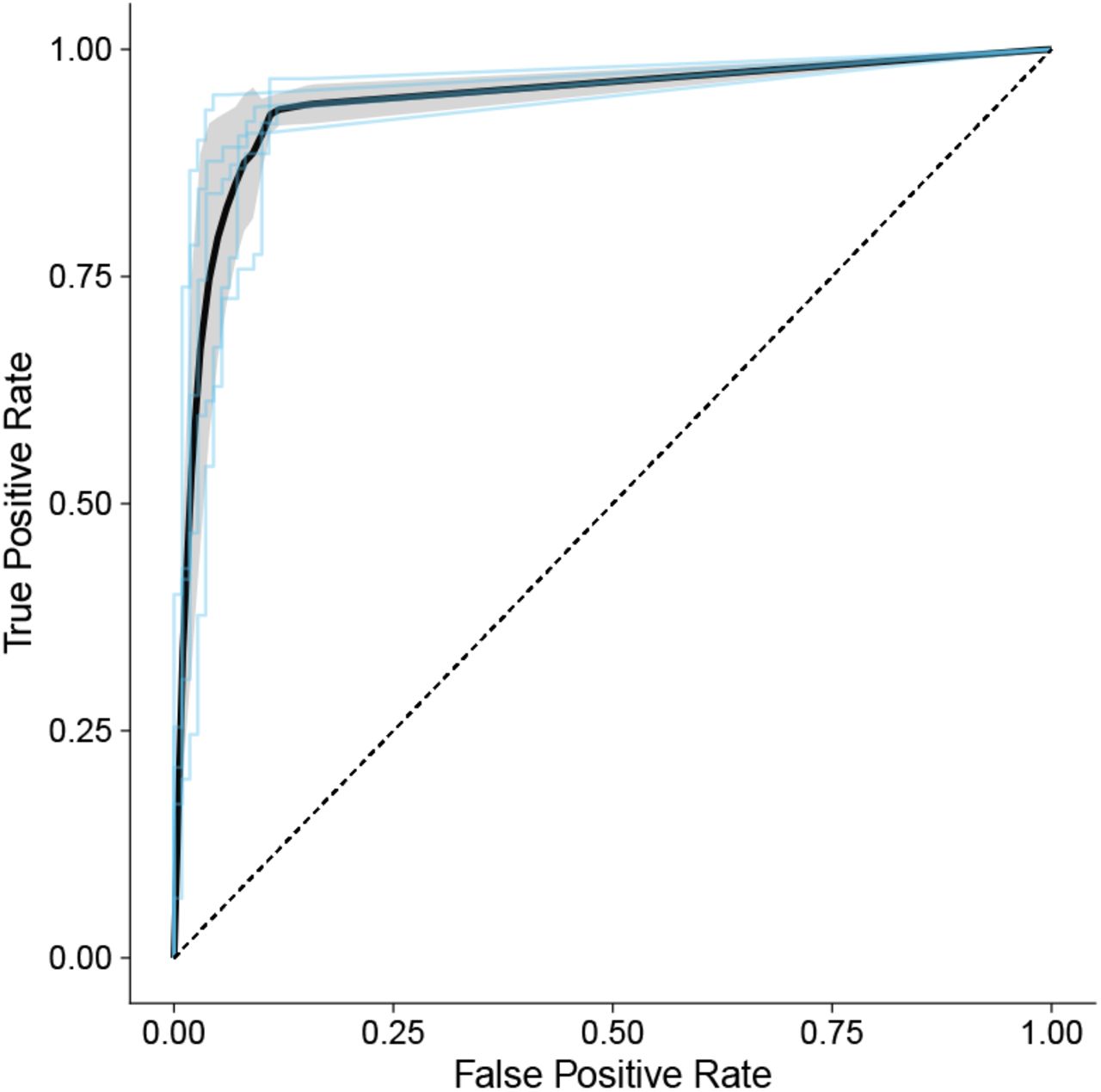
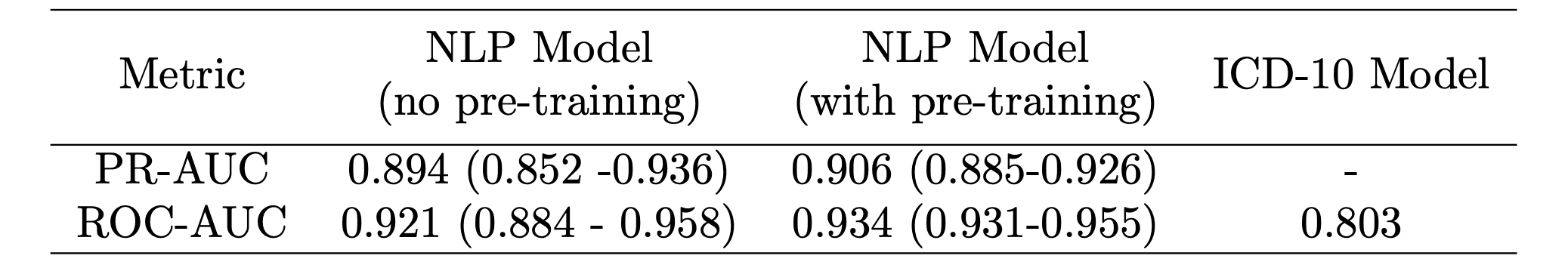
モデルは、ROC-AUC 0.934(95%CI:0.931-0.955)(図 3 参照)および PR-AUC 0.906(95%CI:0.885-0.926)で、ドキュメントレベルで優れた識別パフォーマンスを示しました。 ROC に関しては、5 つのフォールド全てで一貫して良好なパフォーマンスが得られました(図 3 参照)。特に最終モデルのパフォーマンスは最高で、 中間の事前学習により PR-AUC が 0.12、ROC-AUC が 0.13 改善され、最終的なモデルの ROC-AUC は、過去に発表された ICD-10 モデルよりも高くなりました(表 1 参照)。
図3 5-分割交差検証によるROC曲線
※平均パフォーマンスは黒い実線で表され、灰色の網掛け部分は平均 ± 標準偏差を表します。
表1 本論文のNLPモデル(事前学習あり/なし)と過去に発表されたICD-10モデルのPR-AUC、ROC-AUCの比較
※事前学習ありの NLP モデルがROC-AUC 0.934、PR-AUC 0.906と最高のパフォーマンスを示した。
NER の出力確率を見ると、モデルは「好酸球」や「発疹」といった適切な単語を一般的な ADR の特徴として正しく認識しています(図 4 参照)。これをみると、多少のスペルミスがあっても堅牢で、該当の単語が ADR クラスの分類に重要度高く寄与していることが分かります。
図4 単語レベルでのADRの確率予測
※カラーグラデーションは、赤(非常に低い確率)から緑(非常に高い確率)に変化し、各単語が ADR クラスに属する確率を表します。
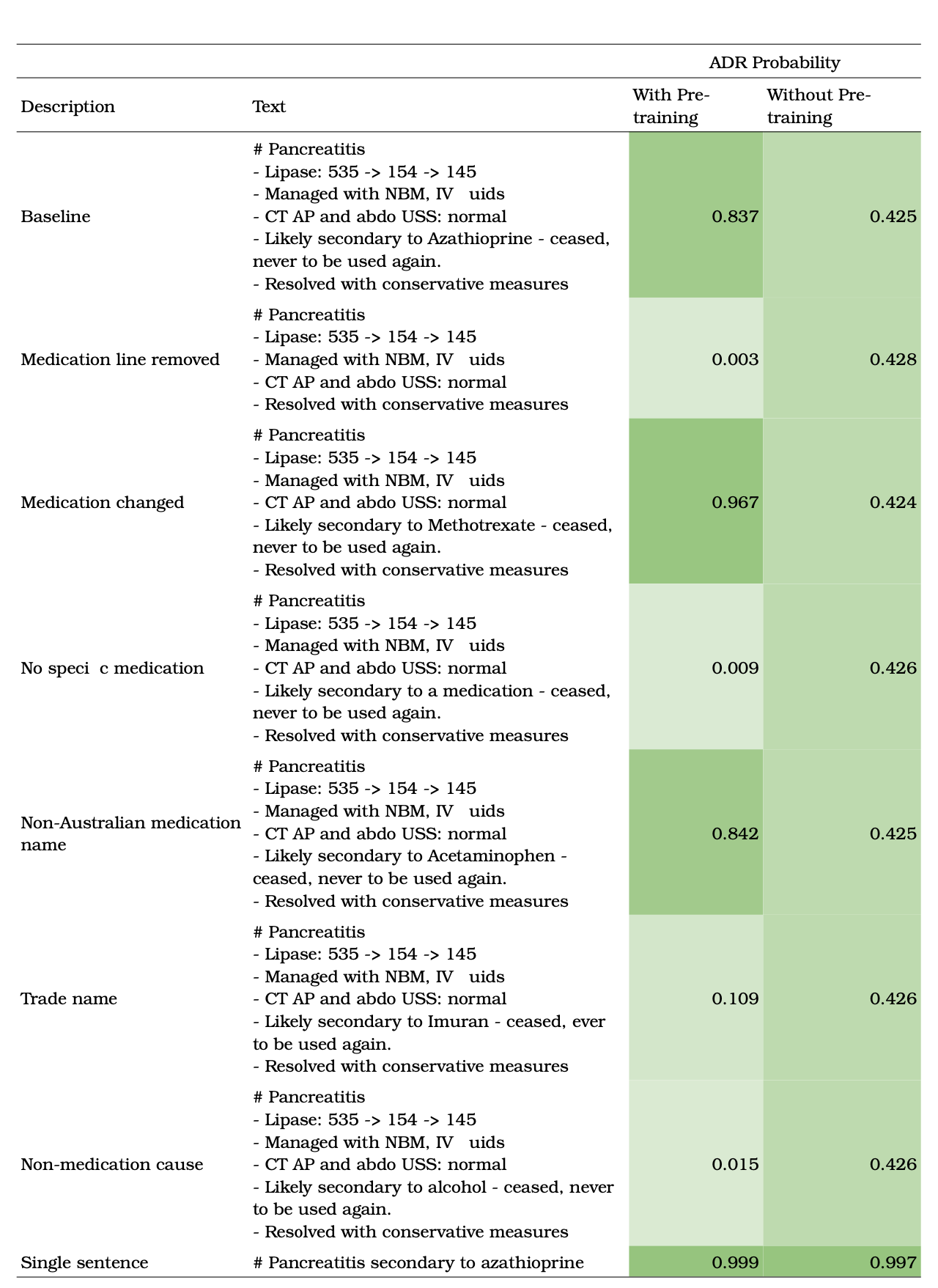
退院サマリーの修正を検証した結果、このアルゴリズムは薬剤の記載に敏感であることが判明しました(表 2 参照)。「アセトアミノフェン」のようなオーストラリア以外の医薬品名には頑健であるように見えますが、商品名は誤分類につながる可能性があります。これは、一般名が多く使用されている著者らの機関での文書化の慣行を反映しているように見えます。薬剤の言及を削除するか、それを病気の寄与や「a medication」などの一般的な用語に置き換えると、予測される ADR の確率が大幅に低下します。事前学習ありのモデルとなしのモデルを比較すると、事前学習ありのモデルは、ADR とその原因となる薬剤の記述が離れている場合でも堅牢な予測を生成します。事前学習を行わないモデルでは、これらの単語がテキスト内で密接に関連している必要があります。
表2 テキストレベルのADR検出率の比較
※事前学習ありのモデルとなしのモデルの ADR 検出率比較。
課題と展望
データセットに関しては、モデルは EMR データの全領域ではなく、ICD-10 でコーディングされた Y40-Y59 の退院サマリーのみを用いてトレーニングされました。ICD-10 コーディングは、臨床医と臨床コーダーが ADE と考えられるイベントを文書化して特定したことに依存しています。このため、コーディングの範囲外にある退院サマリーでは、重要な交絡因子が見落とされている可能性があります。退院サマリー以外にも、他の EMR の記録(例:入院患者の記録、病理報告書など)を包含するようにデータセット を拡張することも今後の研究の方向性の一つです。
参考文献
[5] Prodigy – Radically efficient machine teaching. An annotation tool powered by active learning.